目的:测试:筛选变异系数最大的前3个基因,对所有样本作PCA分析
1
2
3
4
5
6
7
8
9
10
11
12
library(useful)
setwd("~/data/ciona_i/rna_seq/expression/stringtie/ref_only/")
read.table("gene_ftkm_all_samples.tsv",header=TRUE,row.names = 1,sep="\t")->expression_data
expression_data[,1:6]->expression_data
as.matrix(expression_data)->expression_data
#去除表达值全为0的行,有些还会作其它筛选,如低表达,等
expression_data_nonzeron<-expression_data[rowSums(expression_data)!=0,]
#对于表达谱数据,因为涉及到PCR的指数扩增,一般会取log处理
#其它数据log处理会降低数据之间的差异,不一定适用
expression_log2<-log2(expression_data_nonzeron+1)
计算变异系数:
1
cv<-apply(expression_log2,1,sd)/rowMeans(expression_log2)
根据变异系数排序:
1
expression_log2<-expression_log2[order(cv,decreasing = T),]
计算中值绝对偏差 (MAD, median absolute deviation)度量基因表达变化幅度。在基因表达中,尽管某些基因很小的变化会导致重要的生物学意义,但是很小的观察值会引入很大的背景噪音,因此也意义不大。
1
2
mads<-apply(expression_log2,1,mad)
expression_log2<-expression_log2[rev(order(mads)),]
筛选前3行:
1
data_var3<-expression_log2[1:3,]
转置,使得矩阵每一行为一个样品,每一列为变量,即每个基因表达量
1
data_var3_forPCA<-t(data_var3)
1
2
# 获得样品分组信息
sample <- rownames(data_var3_forPCA)
把样品名字按 <_> 分割,取出其第二部分作为样品的组名
1
2
###lapply(X, FUC) 对列表或向量中每个元素执行FUC操作,FUNC为自定义或R自带的函数
group <- unlist(lapply(strsplit(sample, "_"), function(x) x[1]))
1
2
3
4
5
6
7
8
9
#根据分组数目确定颜色变量
colorA <- rainbow(length(unique(group)))
# 根据每个样品的分组信息获取对应的颜色变量
colors <- colorA[as.factor(group)]
# 根据样品分组信息获得legend的颜色
colorl <- colorA[as.factor(unique(group))]
scatterplot3d(data_var3_forPCA[,1:3], color=colors)
legend("top", legend=levels(as.factor(group)), col=colorl, pch=16, xpd=T, horiz=F, ncol=6)
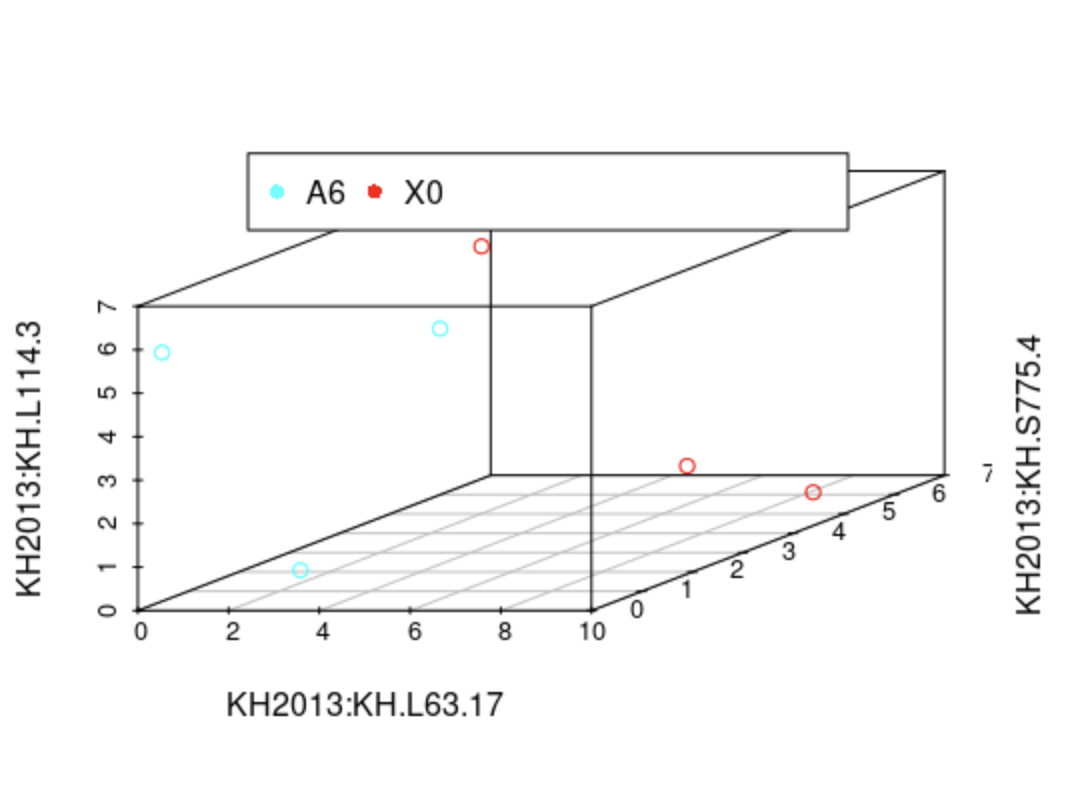
我们看到图中的样品并没有按照预先设定的标签完全分开。当然我们也可以通过其他方法筛选变异最大的三个基因,最终的分类效果不会相差很大。因为不管怎么筛选,我们都只用到了3个基因的表达量。
PCA
假如我们把这个数据用PCA来分类,结果是怎样的呢?
用FPKM
对之前log化的表达矩阵转置:,行为基因,列为样本
1
expression_log2_t<-t(expression_log2)
1
2
3
4
#group在前面定义过了,Add group column for plotting
label<-as.data.frame(expression_log2_t)
label$group<-group
prcomp() 函数默认中心化为减去平均值,标准化用标准差
1
2
3
4
5
6
7
# By default, prcomp will centralized the data using mean.
# Normalize data for PCA by dividing each data by column standard deviation.
# Often, we would normalize data.
# Only when we care about the real number changes other than the trends,
# `scale` can be set to TRUE.
# We will show the differences of scaling and un-scaling effects.
pca<-prcomp(expression_log2_t,scale=T)
1
2
3
# sdev: standard deviation of the principle components.
# Square to get variance
percentVar <- pca$sdev^2 / sum( pca$sdev^2)
1
2
library(ggfortify)
autoplot(pca, data=label, colour="group") + xlab(paste0("PC1 (", round(percentVar[1]*100), "% variance)")) + ylab(paste0("PC2 (", round(percentVar[2]*100), "% variance)")) + theme_bw() + theme(panel.grid.major = element_blank(), panel.grid.minor = element_blank()) + theme(legend.position="right")
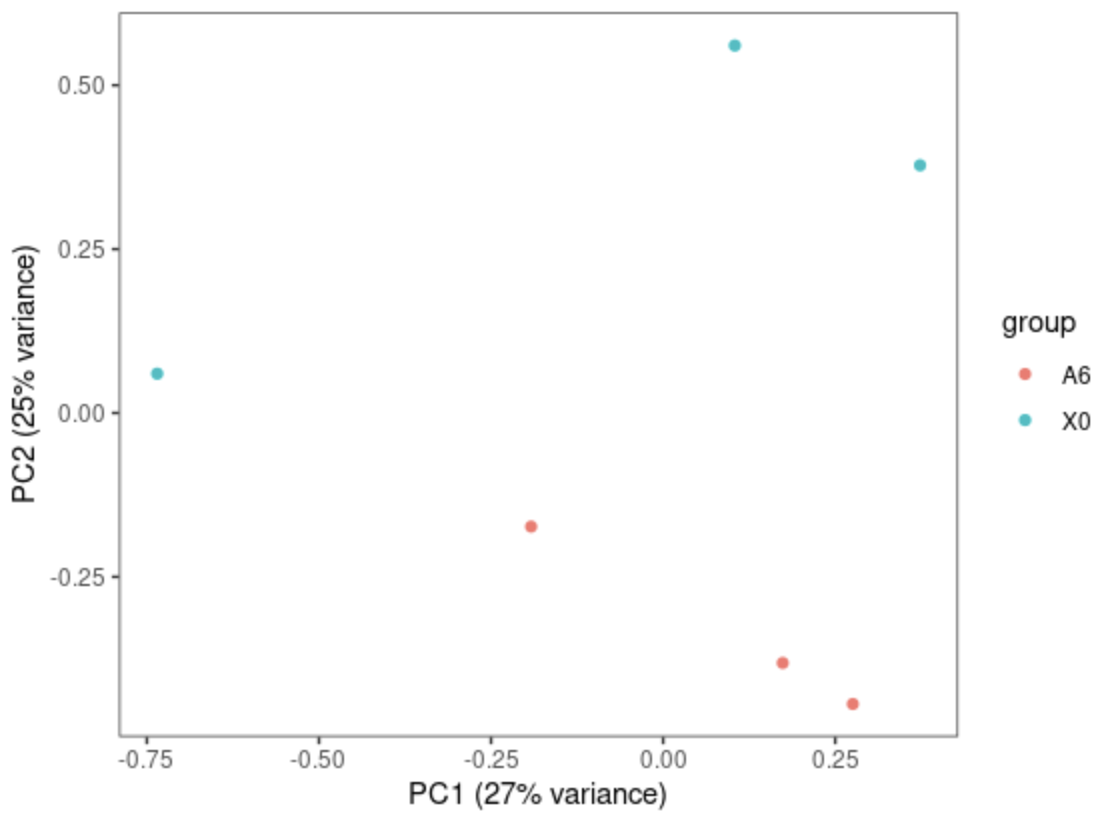
采用3个主成分获得的分类效果优于2个主成分,因为这样保留的原始信息更多。
1
2
3
4
5
6
7
8
9
# 获得PCH symbol列表
pch_l <- as.numeric(as.factor(unique(group)))
# 产生每个样品的pch symbol
pch <- pch_l[as.factor(group)]
pc <- as.data.frame(pca$x)
scatterplot3d(x=pc$PC1, y=pc$PC2, z=pc$PC3, pch=pch, color=colors, xlab=paste0("PC1 (", round(percentVar[1]*100), "% variance)"), ylab=paste0("PC2 (", round(percentVar[2]*100), "% variance)"), zlab=paste0("PC3 (", round(percentVar[3]*100), "% variance)"))
legend(-3,8, legend=levels(as.factor(group)), col=colorl, pch=pch_l, xpd=T, horiz=F, ncol=6)
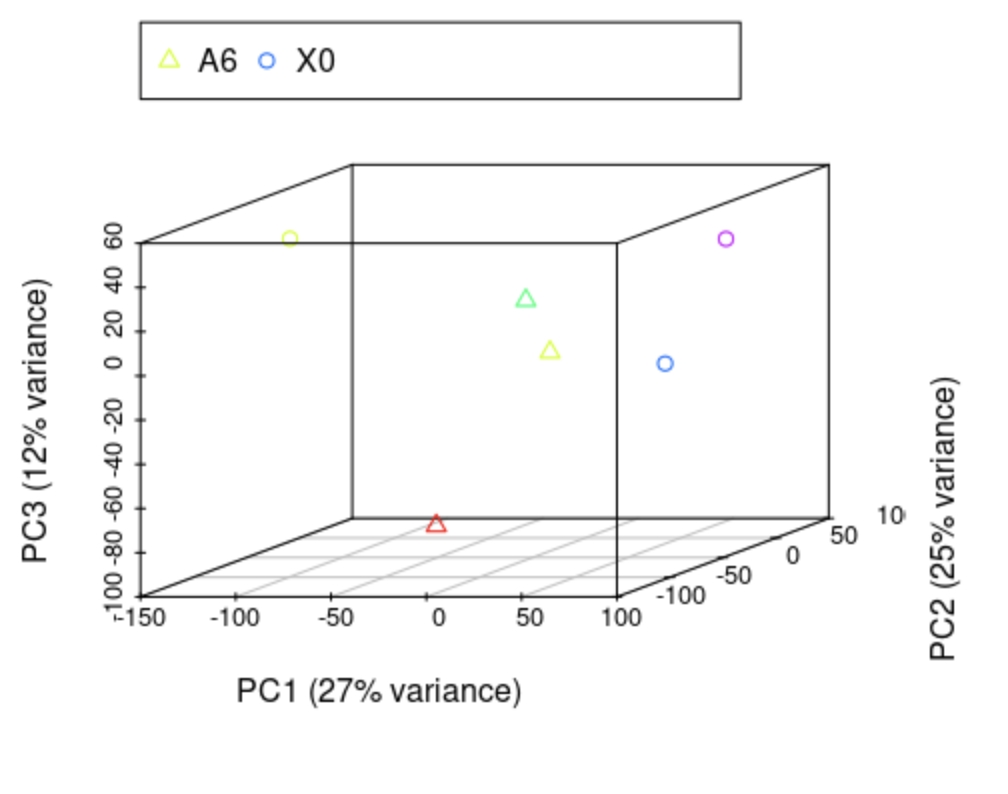
还是没有聚在一起?
当只中心化时:
1
2
3
pca<-prcomp(expression_log2_t)
percentVar <- pca$sdev^2 / sum( pca$sdev^2)
autoplot(pca, data=label, colour="group") + xlab(paste0("PC1 (", round(percentVar[1]*100), "% variance)")) + ylab(paste0("PC2 (", round(percentVar[2]*100), "% variance)")) + theme_bw() + theme(panel.grid.major = element_blank(), panel.grid.minor = element_blank()) + theme(legend.position="right")
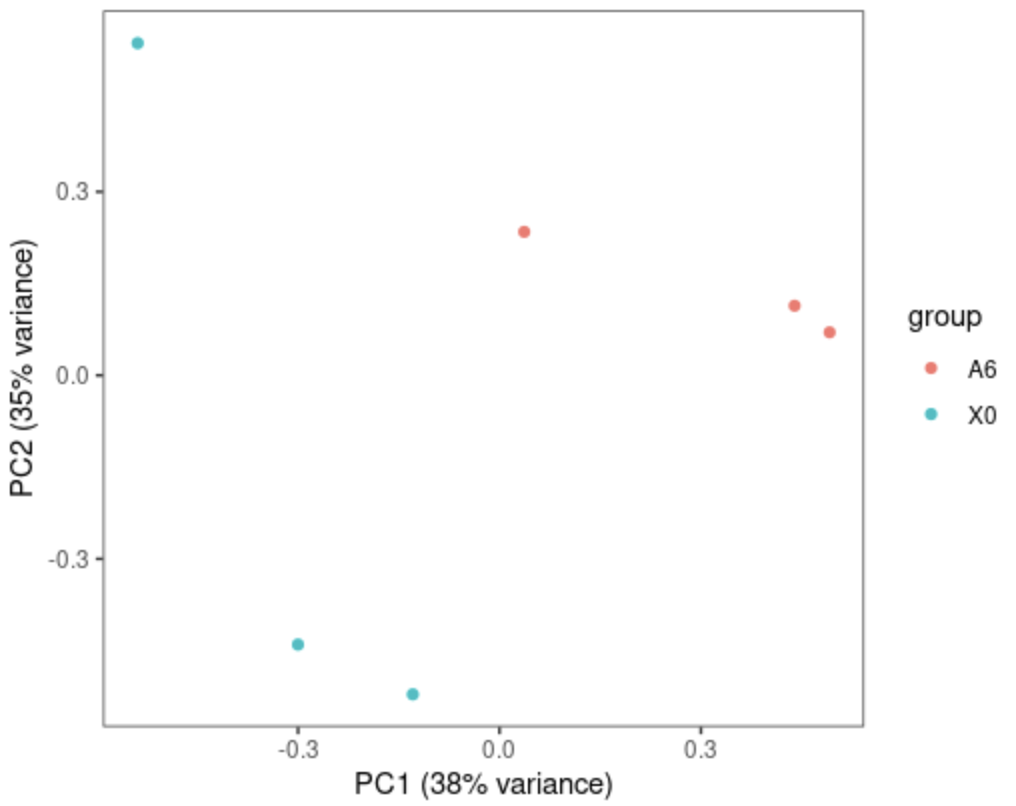
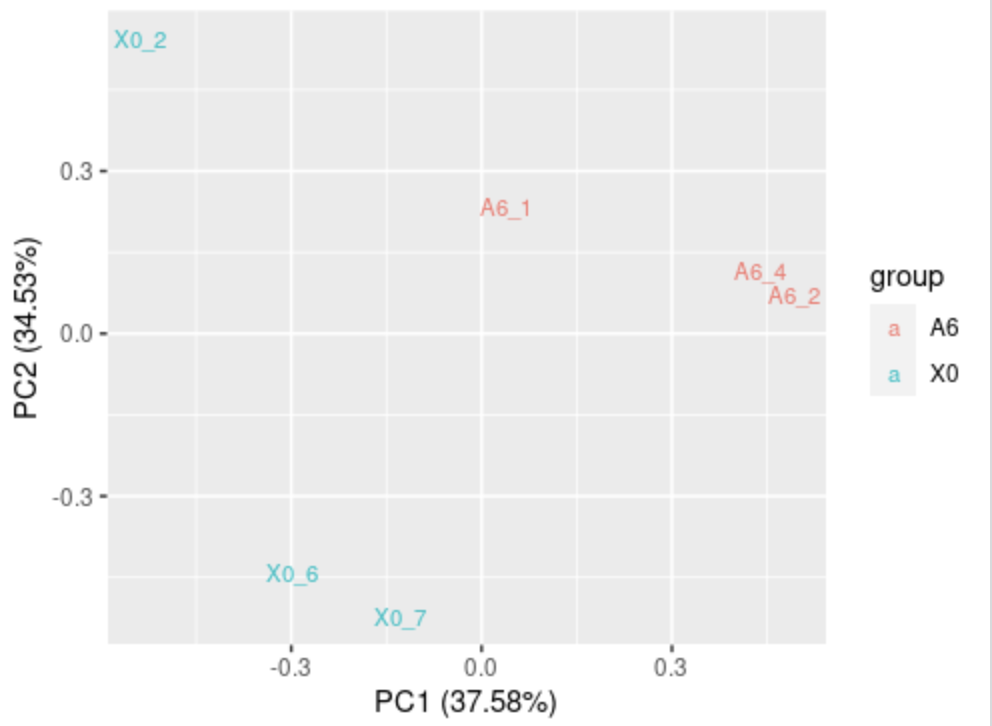
autoplot一行代码自动画:
1
autoplot(prcomp(expression_log2_t),data=label,colour='group',shape=FALSE,label.size=3)
画三维图代码与前面相同:
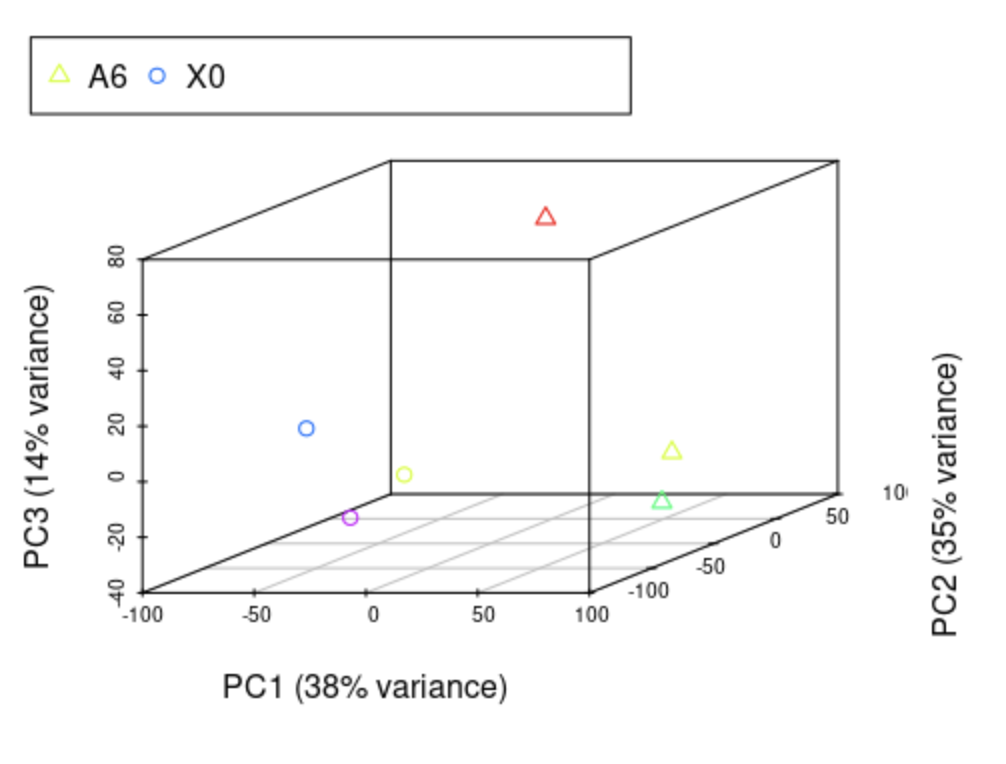
没有聚在一起,看https://www.biostars.org/p/332836/
用raw-count作PCA
归一化参考:https://cloud.tencent.com/developer/article/1486102
有时当表达量为0时,取log会出现错误,可以log(counts+1)来取log值。当x=1时,所有的log系列函数值都为0。这样原本表达量为0的值,取log后仍为0。 这也就是UCSC的XENA下载到的表达矩阵的形式。
读取featureCount结果文件:
1
read.table("all_feature.txt",header=T,skip=1,sep="\t",row.names = 1)->raw_count
取6~11列
1
raw_count[,6:11]->Ci_RawCount
化为矩阵
1
2
3
as.matrix(Ci_RawCount)->Ci_RawCount
##改名字
colnames(Ci_RawCount)<-c("X0_2","X0_7","X0_6","A6_1","A6_2","A6_4")
归一化:
1
2
3
log2(Ci_RawCount+1)->Ci_logCount
##转置
t(Ci_logCount)->Ci_logCount
第一种方法画图:
用ggfortify包画图,遇到“Error: Objects of type prcomp not supported by autoplot.”,重新加载这个包就好了。
1
2
3
4
library(ggfortify)
label_raw<-as.data.frame(Ci_logCount)
label_raw$group<-group
autoplot(prcomp(Ci_logCount),data=label_raw,colour='group',shape=FALSE,label.size=3)
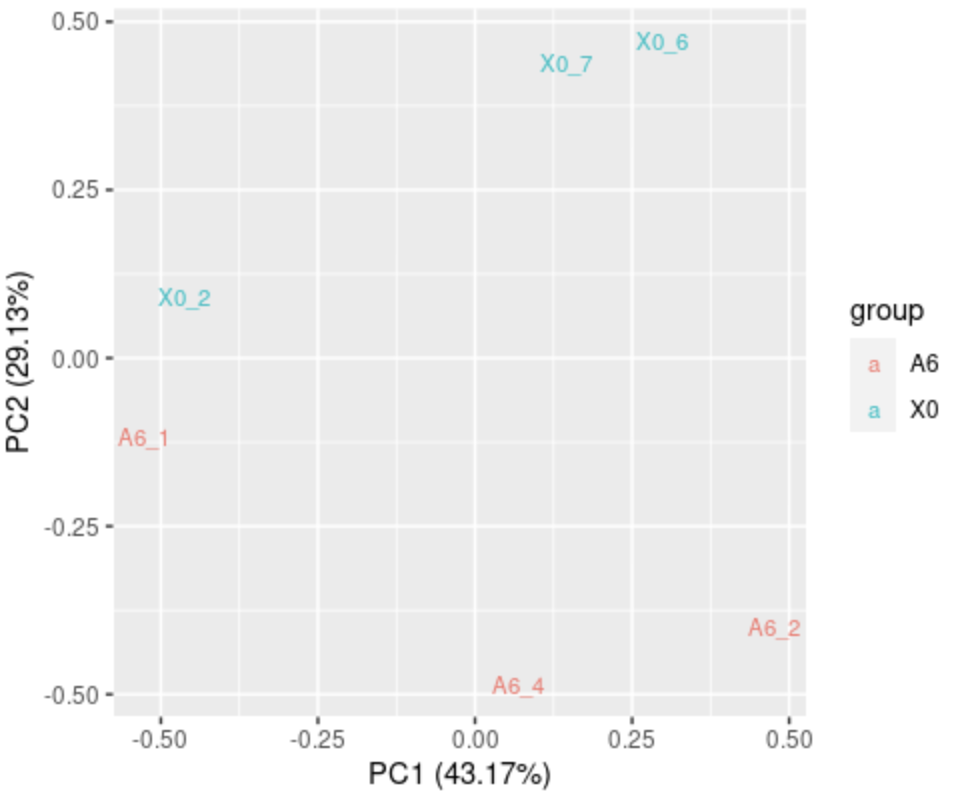
第二种方法画图:
1
2
3
4
5
project.pca <- prcomp(Ci_logCount)
summary(project.pca)
#Determine the proportion of variance of each component
#Proportion of variance equals (PC stdev^2) / (sum all PCs stdev^2)
project.pca.proportionvariances <- ((project.pca$sdev^2) / (sum(project.pca$sdev^2)))*100
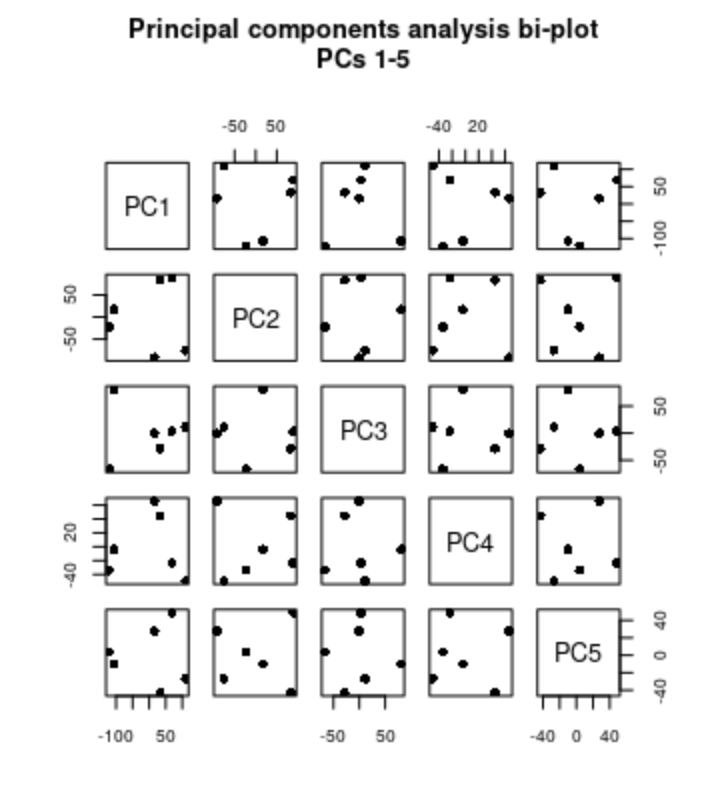
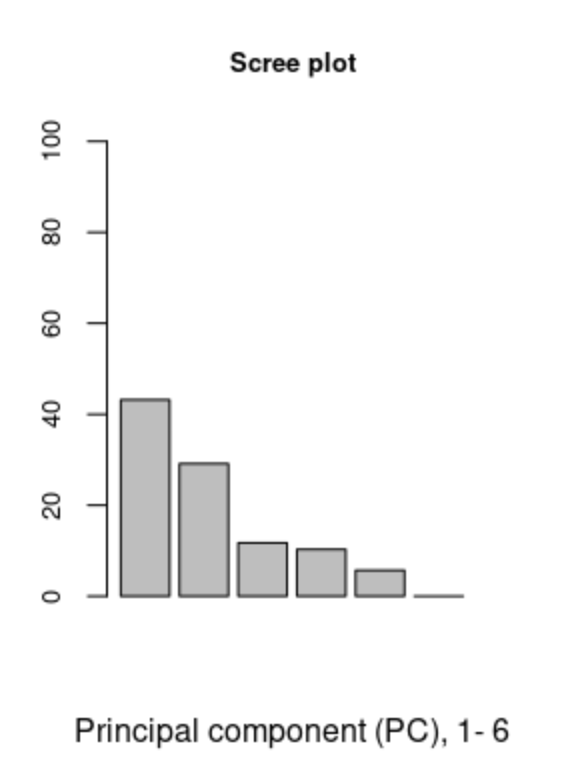
Scree plot
1
barplot(project.pca.proportionvariances, cex.names=1, xlab=paste("Principal component (PC), 1-", length(project.pca$sdev)), ylab="Proportion of variation (%)", main="Scree plot", ylim=c(0,100))
1
2
par(cex=1.0, cex.axis=0.8, cex.main=0.8)
pairs(project.pca$x[,1:5], col="black", main="Principal components analysis bi-plot\nPCs 1-5", pch=16)