教程:
为什么要de novo?:
Genome-guided assembly VS De novo assembly?
1.缺乏高质量参考基因组,
2.RNA-seq 可以注释转录基因和外显子结构
Genomic DNA assembly: one locus-one contig
Transcriptome : one contig per distinct transcript(isoform) rather than per locus;and different transcripts will have different coverage, reflecting their different expression levels.
安装:
1
conda create -n Trinity Trinity -y
使用:
1
Trinity --seqType fq --max_memory 50G --left reads_1.fq --right reads_2.fq --CPU 6 --jaccard_clip
参数说明:
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
21
22
23
24
25
26
27
28
29
30
31
32
33
34
35
36
37
38
39
40
41
42
43
44
45
46
47
48
49
50
51
52
53
54
55
56
57
58
59
60
61
62
63
64
65
66
67
68
必须的参数:
--seqType reads的类型:(cfa, cfq, fa, or fq)
--JM jellyfish使用多少G内存用来进行k-mer的计算,包含‘G’这个字符
--left 左边的reads的文件名
--rigth 右边的reads的文件名
--single 不成对的reads的文件名
可选参数:
Misc:
--SS_lib_type reads的方向。成对的reads: RF or FR; 不成对的reads
: F or R。在数据具有链特异性的时候,设置此参数,则正义和反义转录子能得到区分。默认
情况下,不设置此参数,reads被当作非链特异性处理。FR: 匹配时,read1在5'端上游,
和前导链一致, read2在3'下游, 和前导链反向互补. 或者read2在上游, read1在下游反
向互补; RF: read1在5'端上游, 和前导链反向互补, read2在3'端下游, 和前导链一致;
--output 输出结果文件夹。默认情况下生成trinity_out_dir文件夹并
将输出结果保存到此文件夹中。
--CPU 使用的CPU线程数,默认为2
--min_contig_length 报告出的最短的contig长度。默认为200
--jaccard_clip 如果两个转录子之间有UTR区重叠,则这两个转录子很有可能在
de novo组装的时候被拼接成一条序列,称为融合转录子(Fusion Transcript)。如果有
fastq格式的paired reads,并尽可能减少此类组装错误,则选用此参数。值得说明的是:
1. 适合于基因在基因组比较稠密,转录子经常在UTR区域重叠的物种,比如真菌基因组。而对
于脊椎动物和植物,则不推荐使用此参数; 2. 要求fastq格式的paired reads文件(文件
中reads名分别以/1和/2结尾,以利于软件识别),同时还需要安装bowtie软件用于reads
的比对; 3. 单独使用具有链特异性的RNA-seq数据的时候,能极大地减少UTR重叠区很小的
融合转录子; 4. 此选项耗费运算,若没必要,则不用此参数。
--prep 仅仅准备一些文件(利于I/O)并在kmer计算前停止程序运行
--no_cleanup 保留所有的中间输入文件
--full_cleanup 仅保留Trinity fasta文件,并重命名成${output_dir}.
Trinity.fasta
--cite 显示Trinity文献引证和一些参与的软件工具
--version 报告Trinity版本并推出
Inchworm 和 K-mer 计算相关选项:
--min_kmer_cov 使用Inchworm来计算K-mer数量时候,设置的Kmer的最小值。
默认为1
--inchworm_cpu Inchworm使用的CPU线程数,默认为6和--CPU设置的值中的
小值。
Chrysalis相关选项:
--max_reads_per_graph 在一个Bruijn图中锚定的最大的reads数目,默认为200
000
--no_run_chrysalis 运行Inchworm完毕,在运行chrysalis之前停止运行
Trinity
--no_run_quantifygraph 在平行化运算quantifygrahp前停止运行Trinity
Butterfly相关选项:
--bfly_opts Butterfly额外的参数
--max_number_of_paths_per_node 从node A -> B,最多允许多少条路径。默认
为10
--group_pairs_distance 最大插入片读长度,默认为500
--path_reinforcement_distance 延长转录子路径时候,reads间最小的重叠碱基
数。默认PE:75; SE:25
--no_triplet_lock 不锁定triplet-supported nodes
--bflyHeapSpaceMax 运行Butterfly时java最大的堆积空间,默认
为20G
--bflyHeapSpaceInit java初始的堆积空间,默认为1G
--bflyGCThreads java进行无用信息的整理时使用的线程数,默
认由java来决定
--bflyCPU 运行Butterfly时使用的CPU线程数,默认为2
--bflyCalculateCPU 计算Butterfly所运行的CPU线程数,由公式
80% * max_memory / maxbflyHeapSpaceMax 得到
--no_run_butterfly 在Chrysalis运行完毕后,停止运行Butterfly
Grid-computing选项:
--grid_computing_module 选定Perl模块,在/Users/bhaas/SVN/trinityr
naseq/trunk/PerlLibAdaptors/。
results:
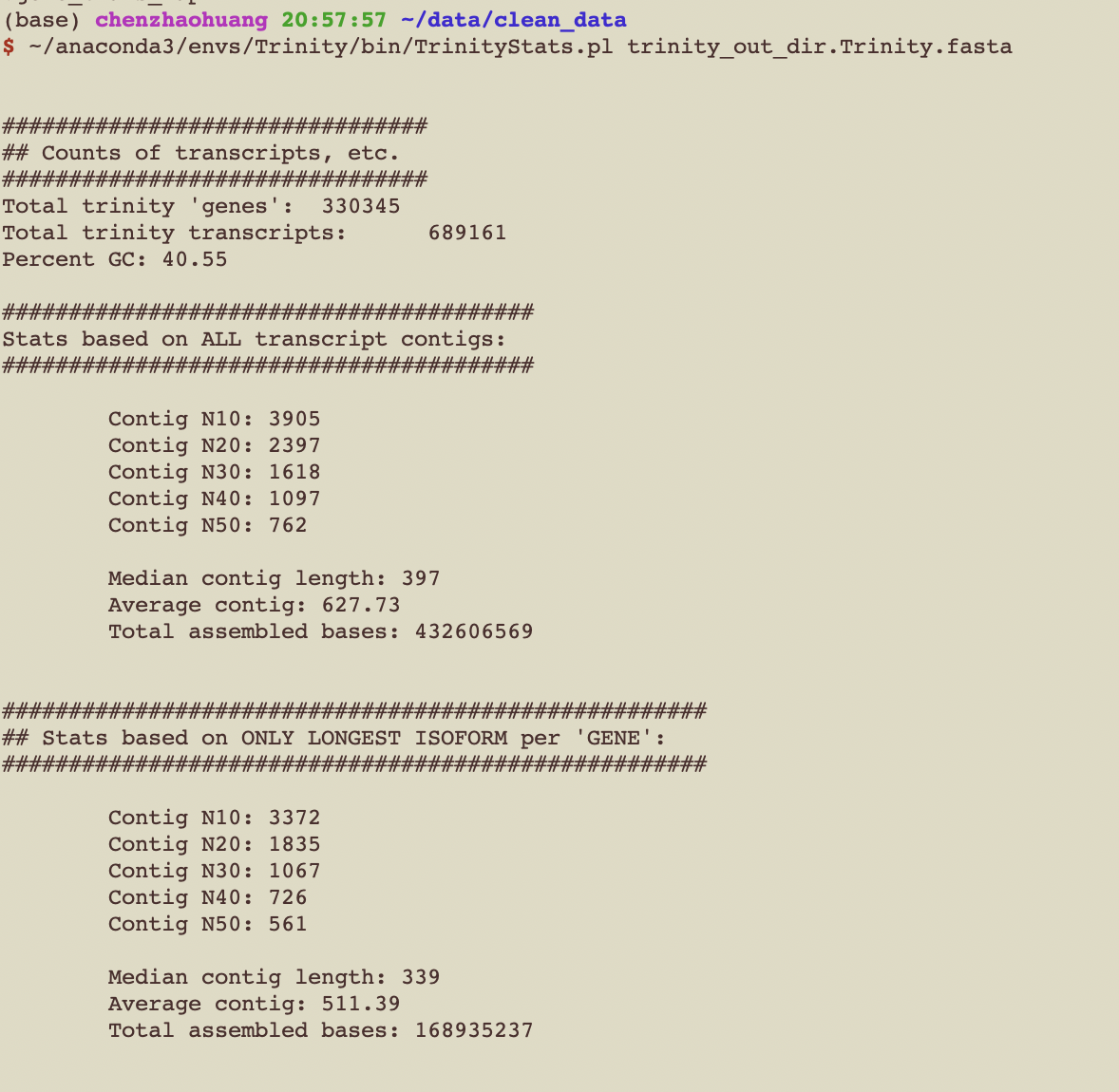
评估组装质量:
1.RNA Seq Read Representation by Trinity Assembly
1
bowtie2-build Trinity.fasta Trinity.fasta
1
bowtie2 -p 30 -q --no-unal -k 20 -x Trinity.fasta -1 ../C1_1_1.fq -2 ../C1_1_2.fq 2>align_stats.txt| samtools view -@30 -Sb -o bowtie2.bam &
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
30251890 reads; of these:
30251890 (100.00%) were paired; of these:
6978680 (23.07%) aligned concordantly 0 times
1325558 (4.38%) aligned concordantly exactly 1 time
21947652 (72.55%) aligned concordantly >1 times
----
6978680 pairs aligned concordantly 0 times; of these:
199258 (2.86%) aligned discordantly 1 time
----
6779422 pairs aligned 0 times concordantly or discordantly; of these:
13558844 mates make up the pairs; of these:
1267412 (9.35%) aligned 0 times
243382 (1.80%) aligned exactly 1 time
12048050 (88.86%) aligned >1 times
97.91% overall alignment rate
比对率大于80%。
2.Full-length transcript analysis for model and non-model organisms using BLAST+
对全长转录本评估可从三方面:
1.align the assembled transcripts to the reference transcripts and examine the length coverage.
2.or non-model organisms, no such reference transcript set is available. If a high quality annotation exists for a closely related organism, then one might compare the assembled transcripts to that closely related transcriptome to examine full-length coverage.
3.In other cases, a more general analysis to perform is to align the assembled transcripts against all known proteins and to determine the number of unique top matching proteins that align across more than X% of its length.
操作:
1.从 ANiseed 下载 C.savignyi 的 Transcript Models:

为什么是protein?
1
服务器文件位置:~/data/clean_data/trinity_out_dir/protein_data
1
2
3
makeblastdb -in Ciona_savignyi.CSAV2.0.81_pep.2018.fa -dbtype prot
blastx -query trinity_out_dir.Trinity.fasta -db protein_data/Ciona_savignyi.CSAV2.0.81_pep.2018.fa -out blastx.outfmt6 -evalue 1e-20 -num_threads 20 -max_target_seqs 1 -outfmt 6
1
analyze_blastPlus_topHit_coverage.pl blastx.outfmt6 trinity_out_dir.Trinity.fasta protein_data/Ciona_savignyi.CSAV2.0.81_pep.2018.fa
结果:
1
2
3
4
5
6
7
8
9
10
11
#hit_pct_cov_bin count_in_bin >bin_below
100 8770 8770
90 815 9585
80 724 10309
70 711 11020
60 741 11761
50 839 12600
40 910 13510
30 1078 14588
20 1317 15905
10 691 16596
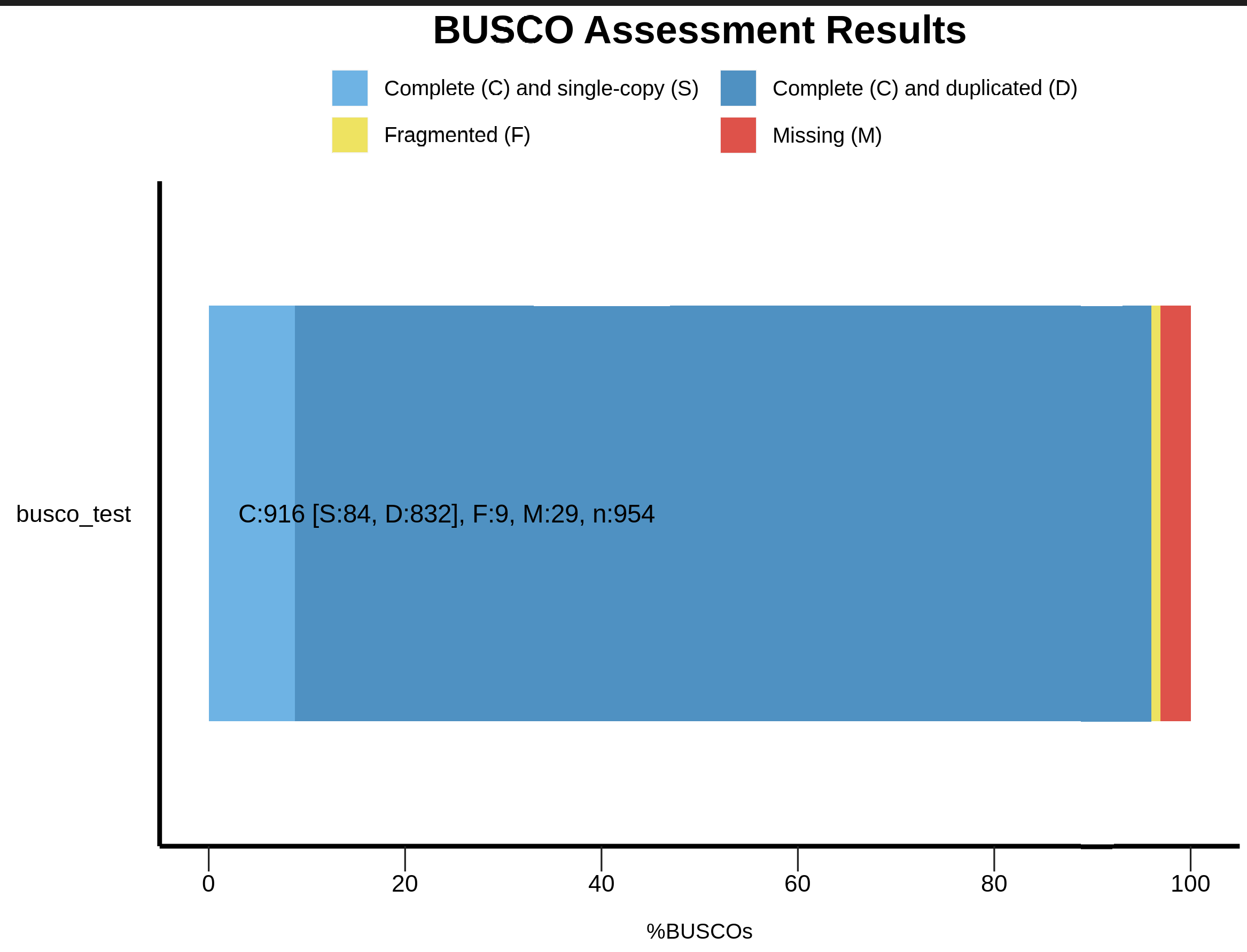
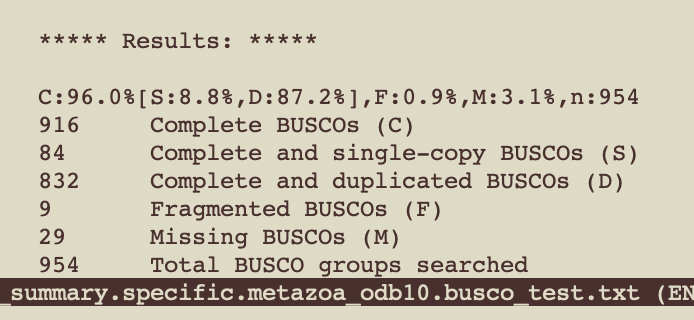
BuSCO
参考:https://busco.ezlab.org/busco_userguide.html
简书:https://www.jianshu.com/p/5041460f7a5d
1
busco -m MODE -i INPUT -o OUTPUT -l LINEAGE

下游分析
基于比对
rsem
1
align_and_estimate_abundance.pl --transcripts trinity_out_dir.Trinity.fasta --seqType fq --samples_file ../samples_trinity.txt --est_method RSEM --output_dir transcript_quant --thread_count 54 --trinity_mode --aln_method bowtie2 --prep_reference